Tema 12 - Bioenergética y oxidación biológica
|
Cap.19: pp.349-351, 353-360
o bien
Cap.10, pp. 220, 226-237
o bien
Cap. 4.3: pp. 115-122
o bien
Cap. 13.1, 13.2, 13.5
(pp. 562-571, 585-587)
|
Introducción al metabolismo.
Termodinámica de los sistemas biológicos. Compuestos ricos en energía.
Oxidorreducción biológica.
|
 Esquemas en Biomodel y Esquemas en Biomodel y
pp.388-401
o bien
Cap.12, pp. 284-288
o bien
Cap. 10.1 y 10.2:
pp. 326-339 y 343 |
Cadena de transporte electrónico.
Fosforilación oxidativa. Estructura y función de la ATP sintasa.
¡Atención!: hay erratas en el texto de McKee.
|
Introducción al metabolismo
pp.12-14 (Presentación de la sección I e introducción del cap. 1)
Producción y utilización de energía en los seres vivos.
Metabolismo: conjunto ordenado de reacciones que se producen en las células
- catalizadas enzimáticamente
- transforman la materia, intercambian energía
- Reacciones encadenadas: vías o rutas metabólicas
- Interdependientes, conectadas
- Su actividad está coordinada: regulación
Metabolismo: anabolismo y catabolismo.
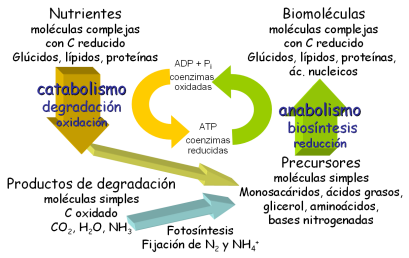
Metabolismo y oxidorreducción:
- Catabolismo es oxidación
- Anabolismo es reducción
- Coenzimas redox: NAD, NADP, FAD, FMN
Bioenergética
Nucleósidos y nucleótidos:
Genérico:
| base nitrogenada |
nucleósido |
nucleótido |
| pentosa |
| fosfato(s) |
|
|
Ejemplo:
| adenina = Ade |
adenosina = Ado |
monofostato de adenosina = adenilato = AMP |
| ribosa = Rib |
| fosfato = P |
|
|
Función del ATP: pp. 355-356
- Es un nucleósido-trifosfato (trifosfato de adenosina)
- base nitrogenada + pentosa = nucleósido
- fosfato(s) + nucleósido = nucleótido
- ej: adenina (Ade) + ribosa (Rib) = adenosina (Ado)
- fostato + adenosina = adenosina-monofosfato (AMP, adenilato)
- difostato + adenosina = adenosina-difosfato (ADP)
- fostato + adenosina = adenosina-trifosfato (ATP)
- Enlaces “ricos en energía” o “de alta energía”: enlaces fosfoanhídrido (anhídrido fosfórico)
- ATP es intermediario, almacén de energía, entre reacciones que liberan (catabolismo) y reacciones que consumen (anabolismo)
“Moneda energética” de los seres vivos.
 Interconversiones del ATP
Interconversiones del ATP
Compuestos “ricos en energía”
fig. 19.7
La ruptura de un enlace (p.ej. por hidrólisis) libera mucha energía libre, al menos 30 kJ/mol
Son termodinámicamente inestables.
| no ricos en energía |
ricos en energía |
- ésteres fosfato: ΔG°' = –4 a –12
Ej.: glucosa-6-fosfato
|
- anhídridos fosfóricos: ΔG°' = –30 a –50. Ej: ATP, ADP
- anhídridos mixtos (fosfórico + carboxílico) Ej.: 1,3-bisfosfoglicerato
- fosfoenoles. Ej.: fosfoenolpiruvato
- tioésteres. Ej: acetil-coenzima A
- fosfoguanidinas. Ej: fosfocreatina
|
 Compuestos ricos en energía (incluye autoevaluación)
Compuestos ricos en energía (incluye autoevaluación)
Reacciones acopladas, obtención y uso de energía
En los seres vivos, las reacciones desfavorables, endergónicas, se hacen posibles “acoplándose” con una reacción favorable, exergónica
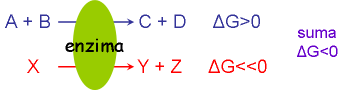 X = compuesto “rico en energía”
X = compuesto “rico en energía”
Los compuestos ricos en energía se obtienen acoplando su formación con reacciones favorables, exergónicas – o bien gracias a la energía de la luz solar
X impulsa reacciones, pero también permite conectar catabolismo con anabolismo.
Ejemplos de acoplamiento energético. Transferencia de fosfato
desde una molécula de alta energía a otra de menor energía. Potencial de transferencia del grupo fosforilo.
Bioenergética:
- Termodinámica
- Energía libre (de Gibbs), G: energía disponible para realizar un trabajo útil
- en una reacción: ΔG (productos – sustratos) = ΔH – T ΔS
- ΔG = 0 equilibrio
- ΔG < 0 reacción exergónica (espontánea)
- ΔG > 0 reacción endergónica (desfavorable)
- ΔG = ΔG° + RT ln ([productos] / [sustratos])
- ΔG° = – RT ln Keq
- En una ruta metabólica, hay que considerar la combinación de las ΔG (unas etapas pueden compensar a otras)
Contenido energético de las moléculas: Valor calórico de las sustancias de los alimentos.
Termodinámica de las reacciones redox. Oxidorreducción biológica
Los procesos metabólicos son redox en buena parte
- Además de intercambio energético hay producción o gasto de “equivalentes de reducción”
- Las coenzimas redox (*) sirven para ello y permiten conectar las reacciones de oxidación (catabolismo) con las reductoras (anabolismo)
- equivalentes redox
 energía química
energía química
- En las reacciones se transfieren 2 protones y 2 electrones
*) estructuras en Biomodel 
Potencial de reducción
- Reducción = ganancia de electrones, A + e−
 A−
A−
- Reductor: tiene capacidad para ceder sus electrones a otra molécula, para ello se oxida (cede electrones)
- Puesto que la semirreacción de reducción es captación A + e−
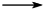 A− ; E⁰ < 0
A− ; E⁰ < 0
el potencial “de reducción” de “A”, para captar electrones, es bajo; valor E⁰ bajo (muy negativo) corresponde a reductores potentes.
- Potencial normal o estándar, E⁰: a 25°C, 1 atm, 1M
E⁰′ es a pH=7 (mejor referencia)
- Para una especie, pareja forma oxidada / forma reducida:
- referencia hidrógeno: E⁰ = 0
H++ e− 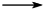 ½ H2
½ H2
- reductores: E⁰ < 0; más reductor: potencial más negativo
- oxidantes: E⁰ > 0 (pero siempre es relativo)
- Ejemplo en una reacción redox:
| |
piruvato + 2H+ + 2e− |
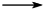 |
lactato |
; E⁰′ = −0.1 V |
| |
NADH |
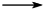 |
NAD + H+ + 2e− |
; E⁰′ = −(−0.32 V) |
| suma: |
piruvato + NADH + H+ |
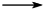 |
lactato + NAD |
; ΔE⁰′ = +0.13 V |
- ΔG⁰ = −n · F · ΔE⁰ (cte. de Faraday F=96.48 kJ·mol−1·V−1) ; ΔE⁰′>0 ; ΔG⁰′<0, reacción espontánea
Flujo o transporte de electrones
Potenciales normales de reducción

- los electrones fluyen, se transfieren...
- desde las especies más reductoras
- hacia las menos reductoras / más oxidantes
- ej.: del NADH al piruvato
- E⁰1 < E⁰2 ; −E1 + E2 = ΔE⁰ >0 ; ΔG⁰ <0, espontánea
La mitocondria
p. 174 ; pp. 65-66 ; cap. 13.5 (pp 585-587)
- Membrana mitocondrial externa
- ~35% proteínas,
~65% lípidos
- Bastante permeable: canales proteicos de porina, transmembranal, poco selectiva, deja pasar <10 kDa
- Espacio intermembranal
- Membrana mitocondrial interna
- ~80% proteínas, ~20% lípidos
- Con crestas (invaginaciones) Muy impermeable; sólo transportadores específicos.
Todas las proteínas del TEM y la ATP sintasa.
- Matriz
- Enzimas del ciclo de Krebs.
- Enzimas de la β-oxidación de ácidos grasos.
- mtDNA, mtRNA, ribosomas
Fosforilación oxidativa
Es la fosforilación del ADP impulsada por el gradiente de protones generado en la cadena de transporte electrónico.
Terminología:
- Teoría de acoplamiento quimiosmótico (Peter Mitchell)
- Gradiente químico (de concentración de H+)
- Gradiente eléctrico.
- Gradiente electroquímico.
- “Fuerza protón-motriz”
Evidencias experimentales.
- Ionóforos y desacoplantes. p.400
- Desacoplamiento fisiológico: termogenina (UCP1) en la grasa parda. p.400
ATP sintasa
pp. 355-356 pero lo desarrollamos completo en Biomodel: 
Modelo quimiosmótico: el gradiente de protones impulsa la acción de la ATP sintasa, que sintetiza ATP fosforilando el ADP con Pi.
Conceptos más importantes de la fosforilación oxidativa:
- Estructura de la ATP sintasa:
- complejo Fo, insertado en la membrana, giratorio (aprox.)
- complejo F1, expuesto en la matriz, estático (aprox.)
- Descripción de las subunidades:
-
| subunidades |
forma parte de |
carácter |
ubicación |
movimiento |
| c12 "carrusel" |
Fo |
hidrófobo |
membrana |
giratorio |
| (α β)3 |
F1 |
hidrófilo |
matriz |
estático |
| γ, ε, δ
"eje" |
¿? |
|
|
giratorio |
| a |
( Fo ) |
hidrófobo |
membrana |
estático |
| b2 |
¿? |
|
ambas |
estático |
Nota: en la ATP sintasa de mitocondrias de mamíferos, la subunidad delta forma parte del eje junto con gamma y épsilon (así se muestra aquí y en los esquemas de Biomodel). En algunos libros se muestra asociada a las subunidades b y al hexámero alfa-beta, pero eso es otra subunidad delta diferente, que forma parte de la ATP sintasa de bacterias. (Referencia)
- Descripción del mecanismo:
- Los protones entran por “a”; para poder salir al lado de la matriz deben asociarse a las “c” y éstas deben girar. (Consulta la nota sobre erratas en McKee)
El flujo de protones a favor de gradiente (del espacio intermembranal hacia la matriz) hace girar el carrusel de subunidades “c”.
Se produce aprox. un giro completo por cada 12 H+ translocados.
- El “eje” γδε gira junto con el carrusel y “choca” con las (αβ)3 provocando cambios conformacionales que activan la actividad catalítica = síntesis del ATP = fosforilación.
Un giro completo activa sucesivamente los 3 dímeros αβ generando 3 ATP.
- El rendimiento (aprox.) es de 1 ATP por cada 4 protones que pasan.
Más información (subunidades componentes, papel de cada una, mecanismo) en  Biomodel > Metabolismo
Biomodel > Metabolismo
Control de la fosforilación oxidativa
Permite adaptación a la demanda de energía. p. 343
Cadena de transporte electrónico mitocondrial
Conceptos más importantes del transporte electrónico mitocondrial:
- Los transportadores son
- complejos proteicos integrales en la membrana (I, II, III, IV), cada uno formado por varias subunidades
- contienen varios grupos prostéticos redox
- un transportador soluble en la bicapa: la ubiquinona o coenzima Q (un lípido)
- un transportador soluble en agua: el citocromo c (una proteína)
- Dos rutas para el transporte de electrones:
- (metabolitos
 ) NADH
) NADH  cI
cI  Q
Q
- metabolitos
 {FADH2 · cII}
{FADH2 · cII}  Q
Q
- final común: Q
 cIII
cIII  cit.c
cit.c  cIV
cIV  O2
O2
- dentro de cada complejo hay un transporte de electrones entre sus cofactores redox
- Resumen:

- El transporte de electrones (a lo largo de la membrana) va acompañado de una translocación de protones, o transporte a través de la membrana; salen protones de la matriz mitocondrial al espacio intermembranal.
- El transporte tiene lugar en orden creciente de potencial de reducción (de más reductor ΔE⁰<<0, a más oxidante, ΔE⁰>>0)
Información del conjunto y de cada complejo: en  Biomodel > Metabolismo
Biomodel > Metabolismo
Complejo I
Complejo NADH deshidrogenasa
- Sustratos: NADH y
ubiquinona (UQ)
- Productos:
NAD y ubiquinol (UQH2)
- “Bombeo” (transporte, translocación) de 4 H+ hacia fuera (por cada 2 e− transportados)
- Centros Fe-S, núcleos Fe-S, centros ferrosulfurados
- Ubiquinona, UQ, Coenzima Q, CoQ
Complejo II y equivalentes
Complejo succinato deshidrogenasa
- Sustratos:
succinato (u otros metabolitos) y
ubiquinona (UQ)
- Sustrato intermedio: FADH2
- Productos:
fumarato (u otros) y
ubiquinol (UQH2)
- Producto intermedio: FAD
- No hay “bombeo” de H+
Complejo deshidrogenasa de acil-CoA
Deshidrogenasa de glicerol-3-fosfato
Complejo III
Complejo del citocromo bc1
- Sustratos:
ubiquinol (UQH2) y
citocromo c{FeIII}
- Productos:
ubiquinona (UQ) y
citocromo c{FeII}
- Bombeo” (transporte, translocación) de 2+2 H+ hacia fuera (por cada 2 e− transportados) (atención: errata, antes decía 2 H+)
- “Ciclo Q” es complejo, y no lo estudiaremos.
- Citocromos: hemo{FeIII} + e−
 hemo{FeII}
hemo{FeII}
Complejo IV
Complejo citocromo oxidasa
- Sustratos:
citocromo c{FeII} y
O2
- Productos:
citocromo c{FeIII} y
H2O
- “Bombeo” (transporte, translocación) de 2 H+ hacia fuera (por cada 2 e− transportados) (atención: errata, antes decía 4 H+)
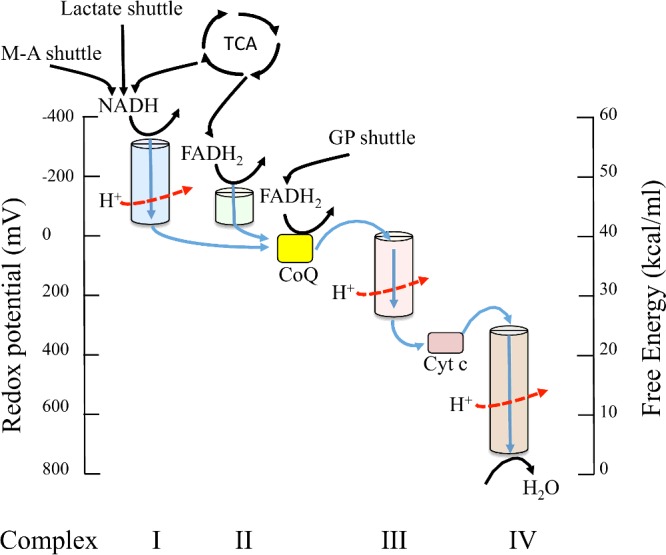
Resumen: potencial redox y energía libre en la cadena de transporte de electrones
- fig. 10.9
- Potencial redox creciente
- Del más reductor al menos reductor
- 3 descensos importantes: 3 aprovechamientos de energía libre
|
licencia CC-by; Ballard & Youngson, doi:10.1042/BSR20150232
Leyenda:
- Lactate shuttle = lanzadera de lactato
- M-A shuttle = lanzadera de malato y aspartato
- TCA = ciclo de ácidos tricarboxílicos (ciclo de Krebs)
- GP shuttle = lanzadera de glicerol-fosfato
|
Inhibición del TEM
- Amital y rotenona inhiben al complejo I
- Antimicina A inhibe al citocromo b en el complejo III
- CO , N3− (azida) y CN− inhiben al complejo IV

Efecto de los inhibidores: ¿qué formas de los transportadores se acumulan?
Obtención de energía a partir de los equivalentes de reducción (resumen)
- En el interior de la mitocondria, el NADH y el FADH2 producidos en el catabolismo (oxidación de los nutrientes) van a conducir a la obtención de energía.
- Se da un proceso de “transporte electrónico” mediante una cadena de reacciones de reducción que tienen lugar en complejos proteicos insertados en la membrana mitocondrial interna

- a la vez se produce un transporte de protones, generando un gradiente entre el interior de la mitocondria y el espacio intermembranal.
- Dicho gradiente de protones impulsa ("fuerza protón-motriz") la fosforilación del ADP (fosforilación oxidativa)
Enfermedades ocasionadas por fallo en la fosforilación oxidativa
pp. 396-399
Mutaciones en DNA mitocondrial
- Codifica 13 subunidades que forman parte de los complejos I, III y IV o del dominio Fo de la ATP sintasa
- Ccodifica los RNA ribosómicos y transferentes mitocondriales, necesarios para la síntesis de las proteínas (traducción)
- Patrón de herencia materna
- La enfermedad suele empeorar con la edad (acumulación de mutaciones en mtDNA por los radicales libres)
- Los síntomas son más severos en en los tejidos con mayor demanda de ATP: nervioso, cardiaco, muscular y renal
Mutaciones en DNA nuclear
- Ccodifica el resto de subunidades que forman parte de los complejos I, II, III y IV o de la ATP sintasa y los transportadores a través de la membrana
- Patrón de herencia normal (paterna y materna): suele ser autosómica recesiva
- Los síntomas son más severos en los tejidos con mayor demanda de ATP
Erratas en el texto de McKee 5ª ed.:
página
impresa |
página del
libro electrónico |
dice |
debe decir |
320 |
337 |
La ATP sintasa consta de dos rotores unidos entre sí por un estator |
La ATP sintasa consta de un rotor en contacto con un estátor |
321 |
338 |
Los protones entran en el anillo c a través de un canal en la subunidad α
(y luego se menciona otras 3 veces) |
la subunidad a
(las 4 veces) |
-(fin)-

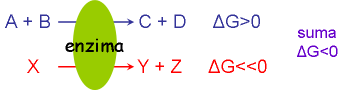 X = compuesto “rico en energía”
X = compuesto “rico en energía”

